機械学習研究者&エンジニアが頭を抱える実験管理に役立つツールを比較した
皆さんこんにちは。
お元気でしょうか。GoogleQA20thで悔しいけど楽しかったです。
自然言語処理のみのコンペを真面目に挑んだのは初で、勉強になることが多かったです。
今回は実験管理ツールの紹介と比較をします。
特徴がわかる範囲で簡単に実装も書いているので、参考にしてみてください。
実験管理ツール
実験管理の必要性
コンペティションや研究では多くのハイパーパラメータや構造などに対して様々な変更を加えます。
私の場合の例ですが、コンペティションだと少なくとも100件ほどは実験します。
しかし、終盤になると実験した内容について忘れてしまい、あれこのハイパーパラメータのときの結果はなんだっけということになりかねません。
今回ご紹介するのはこちらです。
- Excel
- mag
- Weights and Biases
- MLFlow
Excelはツールなのか怪しいのですが、手動記録・BIとしてのベンチマークとして掲載しています。
自動記録以外は案外良いのでありな気もしています。
実験管理ツールの要件
実験管理に必要なものはなんでしょうか?個人的にはこのあたりです。
1. ハイパーパラメータの保存・・・実験した際にハイパーパラメータが記録として残せるか。
2. 学習曲線・・・ニューラルネットワークがメインですが、グラフとしての記述が可能か。
3. 出力物の保存・・・実験結果としてモデルやログなどが残せるか
4. 結果の可視化のリッチ度・・・その得られたものの結果が見やすいか
5. 集約しやすいか・・・リモートサーバにある場合に、1-4の保存が一つのUIなどで見れるかどうか。
実験管理ツールの紹介
Excel
Excelとは
Excelは言わずもがな、Microsoftが開発した表計算ソフトです。
Kaggleでは、ある種、最強のテーブルデータ解析ツールです。
データが少なければJupyter Notebookも必要なく、カラムのフィルタやグラフの作成も可能です。
ユーザが直感的に操作できる点もあるため、状況によってはJupyter Notebookよりも便利で、非常に汎用性の高いツールとなります。
全ての記録をExcelで残すには、全て手を動かし、ログの表を書いたり、出力物は紐付けるように保存したりなど、工夫が必要です。
良い点
1. Excelの機能が使える
Excelの機能をフル活用できます。
テーブルに対してフィルタをかける、表をかけるなど自由に記述が可能です。
欠点
1. 自動化が困難
自動化には、非常に困難な壁が立ちはだかっています。
Excelを解析するスクリプトを書いてしまえば、できるかもしれませんが、そこに時間をかけたいエンジニア・研究者はいないでしょう。
大量実験をする際には不便です。
mag
magとは
magはとても簡単で便利な実験管理ツールです。
GoogleQA 1stの方が利用されているのを見て、使ってみたくなりました。
導入までが早く、全てコンソールで実行可能なのは特徴です。
インストールには、次のコマンドを実行してください。
pip install git+https://github.com/ex4sperans/mag.git
サンプル実装
サンプルでirisで実行するならば、次のとおりです。(サンプルまま)
import argparse import os import pickle from sklearn.datasets import load_iris from sklearn.svm import SVC from sklearn.model_selection import cross_val_score, StratifiedKFold from mag.experiment import Experiment parser = argparse.ArgumentParser( formatter_class=argparse.ArgumentDefaultsHelpFormatter) parser.add_argument( "--C", type=float, default=1.0, help="Regularization parameter for SVM") parser.add_argument( "--gamma", type=float, default=0.01, help="Kernel parameter for SVM") parser.add_argument( "--cv", type=int, default=5, help="Number of folds for cross-validation") parser.add_argument( "--cv_random_seed", type=int, default=42, help="Random seed for cross-validation iterator") args = parser.parse_args() svm_config = { "model": { "C": args.C, "gamma": args.gamma }, "crossval": { "n_folds": args.cv, "_random_seed": args.cv_random_seed } } # ハイパーパラメータの定義(1) iris = load_iris() with Experiment(config=svm_config) as experiment: # ここからmag実験履歴の取得を開始 config = experiment.config model = SVC(C=config.model.C, gamma=config.model.gamma) score = cross_val_score( model, X=iris.data, y=iris.target, scoring="accuracy", cv=StratifiedKFold( config.crossval.n_folds, shuffle=True, random_state=config.crossval._random_seed), ).mean() print("Accuracy is", round(score, 4)) experiment.register_result("accuracy", score) # magのスコアの登録をする。 with open(os.path.join(experiment.experiment_dir, "model.pkl"), "wb") as f: # 実験ディレクトリの中で、ファイルを保存する。(2) pickle.dump(model, f)
出力ディレクトリは次の通りです。
experiments/
└── 5-1.0-0.01
├── command
├── config.json
├── log
├── model.pkl
└── results.json特定のディレクトリの中で実験ごとにディレクトリが生成されます。
1. command・・・実行コマンド
2. config.json・・・ハイパーパラメータ(今回だとSVC, (1)に該当する部分)
3. log・・・標準出力の結果
4. model.pkl・・・学習済モデル
5. results.json・・・register_resultで残った記録
また、複数の実験を集約し、比較するためのコマンドも用意されています。
$ python -m mag.summarize experiments --metrics=accuracy
Results for <directory>:
accuracy
5-10.0-1.0 0.953333
5-1.0-0.01 0.933333
良い点
1. 軽量・お手軽
コマンドラインで完結するため、簡単に実験を管理したい場合にちょうど良い塩梅です。
2. コマンド・標準出力の自動保存
実行コマンドや標準出力を勝手に拾ってきてくれるので、諸々printしておけば少なくとも消滅することはありません。
いざ、なにか残っていると後追いでログを確認できますが、それすらもないとどうしようもありません。
ここが少し残念
1. 一覧化できるUIとその機能
標準出力のUIがあまりイケていないため、少し見づらいと感じました。
ソート等も自分でしなければならないのは苦しいですね。
2. 複数サーバの運用は難しそう
ログがローカルに出力されるので、複数台ある場合は統合する仕組みが必要でしょう。
Weights and Biases
Weights and Biasesとは
実験の分析や運用をするためのツールです。
ローカルのクライアントツールを使い、サーバへデータを送信します。
クライアントツールは次のサイトから導入できます。
Webサイトはこちらです。UIで実験結果をこちらで確認できます。
サンプル実装
事前に利用にはコマンドラインからログインが必要です。次のコマンドを実行し、順を追って進めましょう。
wandb login
サンプルコードとして以下のものを用意しました。
import argparse import os import tempfile import torch import torch.nn as nn import torch.nn.functional as F import torch.optim as optim from torchvision import datasets, transforms from torch.autograd import Variable import wandb # Command-line arguments parser = argparse.ArgumentParser(description='PyTorch MNIST Example') parser.add_argument('--batch-size', type=int, default=64, metavar='N', help='input batch size for training (default: 64)') parser.add_argument('--test-batch-size', type=int, default=1000, metavar='N', help='input batch size for testing (default: 1000)') parser.add_argument('--epochs', type=int, default=10, metavar='N', help='number of epochs to train (default: 10)') parser.add_argument('--lr', type=float, default=0.01, metavar='LR', help='learning rate (default: 0.01)') parser.add_argument('--momentum', type=float, default=0.5, metavar='M', help='SGD momentum (default: 0.5)') parser.add_argument('--enable-cuda', type=str, choices=['True', 'False'], default='True', help='enables or disables CUDA training') parser.add_argument('--seed', type=int, default=1, metavar='S', help='random seed (default: 1)') parser.add_argument('--log-interval', type=int, default=100, metavar='N', help='how many batches to wait before logging training status') args = parser.parse_args() enable_cuda_flag = True if args.enable_cuda == 'True' else False args.cuda = enable_cuda_flag and torch.cuda.is_available() torch.manual_seed(args.seed) if args.cuda: torch.cuda.manual_seed(args.seed) kwargs = {'num_workers': 1, 'pin_memory': True} if args.cuda else {} train_loader = torch.utils.data.DataLoader( datasets.MNIST('../data', train=True, download=True, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.1307,), (0.3081,)) ])), batch_size=args.batch_size, shuffle=True, **kwargs) test_loader = torch.utils.data.DataLoader( datasets.MNIST('../data', train=False, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.1307,), (0.3081,)) ])), batch_size=args.test_batch_size, shuffle=True, **kwargs) class Net(nn.Module): def __init__(self): super(Net, self).__init__() self.conv1 = nn.Conv2d(1, 10, kernel_size=5) self.conv2 = nn.Conv2d(10, 20, kernel_size=5) self.conv2_drop = nn.Dropout2d() self.fc1 = nn.Linear(320, 50) self.fc2 = nn.Linear(50, 10) def forward(self, x): x = F.relu(F.max_pool2d(self.conv1(x), 2)) x = F.relu(F.max_pool2d(self.conv2_drop(self.conv2(x)), 2)) x = x.view(-1, 320) x = F.relu(self.fc1(x)) x = F.dropout(x, training=self.training) x = self.fc2(x) return F.log_softmax(x, dim=0) model = Net() if args.cuda: model.cuda() optimizer = optim.SGD(model.parameters(), lr=args.lr, momentum=args.momentum) def train(epoch): model.train() for batch_idx, (data, target) in enumerate(train_loader): if args.cuda: data, target = data.cuda(), target.cuda() data, target = Variable(data), Variable(target) optimizer.zero_grad() output = model(data) loss = F.nll_loss(output, target) loss.backward() optimizer.step() if batch_idx % args.log_interval == 0: print('Train Epoch: {} [{}/{} ({:.0f}%)]\tLoss: {:.6f}'.format( epoch, batch_idx * len(data), len(train_loader.dataset), 100. * batch_idx / len(train_loader), loss.data.item())) step = epoch * len(train_loader) + batch_idx wandb.log({'train_loss': loss.data.item()}) def test(epoch): model.eval() test_loss = 0 correct = 0 with torch.no_grad(): for data, target in test_loader: if args.cuda: data, target = data.cuda(), target.cuda() data, target = Variable(data), Variable(target) output = model(data) test_loss += F.nll_loss(output, target, reduction='sum').data.item() # sum up batch loss pred = output.data.max(1)[1] # get the index of the max log-probability correct += pred.eq(target.data).cpu().sum().item() test_loss /= len(test_loader.dataset) test_accuracy = 100.0 * correct / len(test_loader.dataset) print('\nTest set: Average loss: {:.4f}, Accuracy: {}/{} ({:.0f}%)\n'.format( test_loss, correct, len(test_loader.dataset), test_accuracy)) step = (epoch + 1) * len(train_loader) wandb.init(config=args, project="mnist-test") output_dir = dirpath = tempfile.mkdtemp() # Perform the training for epoch in range(1, args.epochs + 1): train(epoch) test(epoch) torch.save(model.state_dict(), os.path.join(wandb.run.dir, 'model.pt'))
次のコマンドで実行します。
python mnist.py
完了後に作成しておいたプロジェクトへ行くと、結果が一覧化されています。
クリックすると詳細画面にも遷移できます。

また、各個別の画面も揃っています。この画面でファイル等も管理されているので、複数台を利用するには良い環境ではないでしょうか。

良い点
1. 機能の追加に容易に対応できる。
UIはライブラリ管理していないので、常に最新の状態のものが利用できます。
ローカルのマシンに管理すると、新機能が追加された場合に、アップグレード操作が必要になります。
2. 複数台での管理が容易
複数台で実験を行っていても、管理が容易です。
ここが少し残念
1. ネットワーク環境がないと厳しい
ネットワークの環境がなければ当たり前ですが、動かせません。
そのため、環境によってはプログラムが利用できなくなります。
2. Standalone版がない
仕事ではクラウドやサービスを利用できない場面が多くあります。
その場合にうっかり利用してしまうと問題になります。
これはどうすることもできないので、その場合にはWeights and Biasesを利用すべきではないでしょう。
2023/8/2更新
最近知ったのですが、2020年10月頃より、Weights and BiasesのStandalone版が公開されているようです。
ぜひ、興味のある人は使ってみてください。
github.com
MLFlow
MLFlowはプラットフォームです。機械学習のデプロイやトラッキング、実装のパッケージングやデプロイなど幅広くサポートしています。
その中ではいくつかの機能があり、主にMLflow Trackingを実験管理に利用している人が増えています。
このツール、最も関心があり、使い方は調べていましたが、実は自分で触ったことがなく、イメージを持てていないツールの一つでした。
そのため、今回はMNISTでそのサンプルを試します。
サンプル実装
公式のサンプル実装に若干手を加えています。(公式だと、TensorBoardへの組み込みもあります)
実装は次の通りです。
import argparse import os import mlflow import tempfile import torch import torch.nn as nn import torch.nn.functional as F import torch.optim as optim from torchvision import datasets, transforms from torch.autograd import Variable # Command-line arguments parser = argparse.ArgumentParser(description='PyTorch MNIST Example') parser.add_argument('--batch-size', type=int, default=64, metavar='N', help='input batch size for training (default: 64)') parser.add_argument('--test-batch-size', type=int, default=1000, metavar='N', help='input batch size for testing (default: 1000)') parser.add_argument('--epochs', type=int, default=10, metavar='N', help='number of epochs to train (default: 10)') parser.add_argument('--lr', type=float, default=0.01, metavar='LR', help='learning rate (default: 0.01)') parser.add_argument('--momentum', type=float, default=0.5, metavar='M', help='SGD momentum (default: 0.5)') parser.add_argument('--enable-cuda', type=str, choices=['True', 'False'], default='True', help='enables or disables CUDA training') parser.add_argument('--seed', type=int, default=1, metavar='S', help='random seed (default: 1)') parser.add_argument('--log-interval', type=int, default=100, metavar='N', help='how many batches to wait before logging training status') args = parser.parse_args() enable_cuda_flag = True if args.enable_cuda == 'True' else False args.cuda = enable_cuda_flag and torch.cuda.is_available() torch.manual_seed(args.seed) if args.cuda: torch.cuda.manual_seed(args.seed) kwargs = {'num_workers': 1, 'pin_memory': True} if args.cuda else {} train_loader = torch.utils.data.DataLoader( datasets.MNIST('../data', train=True, download=True, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.1307,), (0.3081,)) ])), batch_size=args.batch_size, shuffle=True, **kwargs) test_loader = torch.utils.data.DataLoader( datasets.MNIST('../data', train=False, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.1307,), (0.3081,)) ])), batch_size=args.test_batch_size, shuffle=True, **kwargs) class Net(nn.Module): def __init__(self): super(Net, self).__init__() self.conv1 = nn.Conv2d(1, 10, kernel_size=5) self.conv2 = nn.Conv2d(10, 20, kernel_size=5) self.conv2_drop = nn.Dropout2d() self.fc1 = nn.Linear(320, 50) self.fc2 = nn.Linear(50, 10) def forward(self, x): x = F.relu(F.max_pool2d(self.conv1(x), 2)) x = F.relu(F.max_pool2d(self.conv2_drop(self.conv2(x)), 2)) x = x.view(-1, 320) x = F.relu(self.fc1(x)) x = F.dropout(x, training=self.training) x = self.fc2(x) return F.log_softmax(x, dim=0) model = Net() if args.cuda: model.cuda() optimizer = optim.SGD(model.parameters(), lr=args.lr, momentum=args.momentum) def train(epoch): model.train() for batch_idx, (data, target) in enumerate(train_loader): if args.cuda: data, target = data.cuda(), target.cuda() data, target = Variable(data), Variable(target) optimizer.zero_grad() output = model(data) loss = F.nll_loss(output, target) loss.backward() optimizer.step() if batch_idx % args.log_interval == 0: print('Train Epoch: {} [{}/{} ({:.0f}%)]\tLoss: {:.6f}'.format( epoch, batch_idx * len(data), len(train_loader.dataset), 100. * batch_idx / len(train_loader), loss.data.item())) step = epoch * len(train_loader) + batch_idx log_scalar('train_loss', loss.data.item(), step) def test(epoch): model.eval() test_loss = 0 correct = 0 with torch.no_grad(): for data, target in test_loader: if args.cuda: data, target = data.cuda(), target.cuda() data, target = Variable(data), Variable(target) output = model(data) test_loss += F.nll_loss(output, target, reduction='sum').data.item() # sum up batch loss pred = output.data.max(1)[1] # get the index of the max log-probability correct += pred.eq(target.data).cpu().sum().item() test_loss /= len(test_loader.dataset) test_accuracy = 100.0 * correct / len(test_loader.dataset) print('\nTest set: Average loss: {:.4f}, Accuracy: {}/{} ({:.0f}%)\n'.format( test_loss, correct, len(test_loader.dataset), test_accuracy)) step = (epoch + 1) * len(train_loader) log_scalar('test_loss', test_loss, step) log_scalar('test_accuracy', test_accuracy, step) def log_scalar(name, value, step): """Log a scalar value to both MLflow and TensorBoard""" mlflow.log_metric(name, value) with mlflow.start_run(): # Log our parameters into mlflow for key, value in vars(args).items(): mlflow.log_param(key, value) # Create a SummaryWriter to write TensorBoard events locally output_dir = dirpath = tempfile.mkdtemp() # Perform the training for epoch in range(1, args.epochs + 1): train(epoch) test(epoch) # Upload the TensorBoard event logs as a run artifact with tempfile.TemporaryDirectory() as tmp: filename = os.path.join(tmp, "model.bin") torch.save(model.state_dict(), filename) mlflow.log_artifacts(tmp, artifact_path="results")
実行は次のコマンドを実行します。
python mnist.py && python mnist.py --epochs 20 && python mnist.py --batch-size 32 && python mnist.py --momentum 0.9
mlrunsディレクトリが作られます。このディレクトリに、学習した結果やログが残されています。
この情報を可視化する場合は次のコマンドを実行します。
mlflow ui
そして、localhost:5000にアクセスすれば保存されているディレクトリの情報を可視化できます。
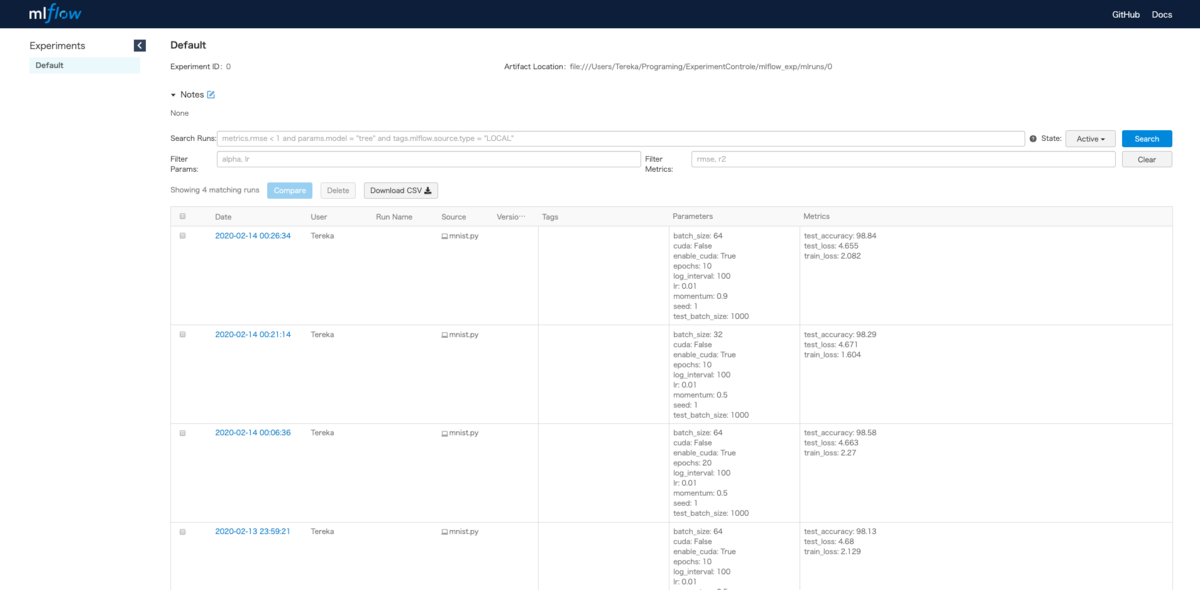
画面を開くと次の画面になり、パラメータとメトリクスが一覧化されて可視化されています。これだけでもかなり見やすい。
フィルタなども記載できるのでおすすめです。このライブラリ相当奥が深そうなので、細かいのは後々紹介します。
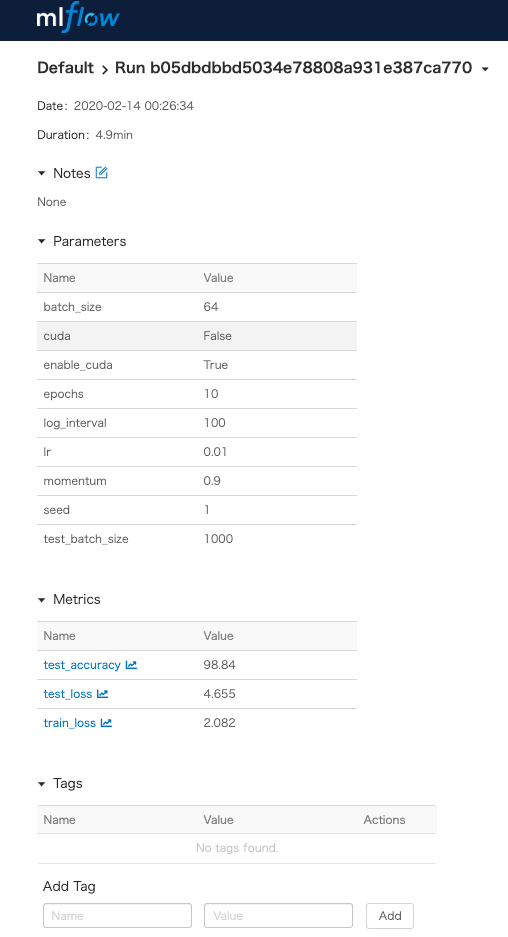
また、各実験の画面を開くと、次のようになります。
今回表示していませんが、この画面で実験の出力物も管理できます。
更にはそのログの遷移を確認できます。
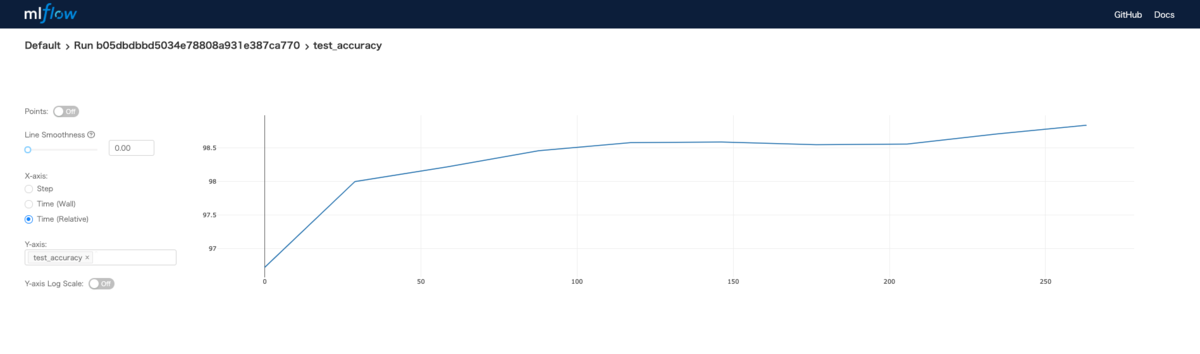
一言言うなれば凄く便利
良い点
1. UIがリッチ
UIがリッチでWeb経由でアクセスできるならば文句ありません。
パラメータのカラムが横ならびになっておらず、各カラムで一覧化されているのは有効でしょう。
2. 多機能
これまでのと比較すると非常に高機能です。
実験において、モデルも併せて記録ができます。また、今回紹介していませんがREST APIも対応しているなど、かなり至れりつくせりです。
3. Standaloneで実行可能
Standaloneで実行できるので、仕事で利用する場合にもうっかりクラウドに情報が漏れ出ることはありません。
wandbにしろ、Comet(他の実験管理)もクラウドに情報が出てしまうのが辛いところです。
ここが少し残念
1. 認証機能
ここまで良いと何人かで共通して使いたいものですが、さすがに、認証機能はありません。
そのため、nginxなど別のOSSを利用して補完する必要があります。
個人で使うには何も問題ありません。
まとめ
正直、MLflow最強では?と思っています。
他のと比較してもどのような環境であっても使える面、UIのリッチさを踏まえると、基本、これを選択すると間違いはないと思っています。
ただ、局所的には他のももちろん良いとも感じています。
magはコンソールで簡単に実施したい場合に有用です。
また、Weights and Biasesはネットワーク環境があれば、Web上から参照でき、かつ、高機能なので選択肢の一つとしてありだとも感じています。
最初の指標に併せて○×つけると次の通りです。MLFlowが万能すぎる。
| 方式 | ハイパーパラメータの保存 | 学習曲線 | 出力物の保存 | 結果の可視化のリッチ度 | 集約しやすいか | 備考 |
|---|---|---|---|---|---|---|
| Excel | ○ | ○ | × | ○ | × | ほぼ手動で頑張る必要あり |
| mag | ○ | × | ○ | × | × | コンソールしかない。 |
| Weights and Biases | ○ | ○ | ○ | ○ | ○ | Standalone版がなく、ネットワーク環境必須 |
| MLFlow | ○ | ○ | ○ | ○ | ○ | 認証機能について、独自実装 or 他OSSが必要 |
最後に
これを書く一つのモチベーションとして実験管理のライブラリは多種多様ですが、何が良いか頭を悩ませていました。
そのため、私の自分のコンペライブラリの中に、ツールをまだ組み込むことは手が動かず、正直できていませんでした。
コンペの合間にようやく、自分の手で動かし、確認できました。少しずつで良いので組み込んでみようと感じています。